-
Humanitaarteaduste ja kunstide valdkondJakobi 2, r 116-121 51005 Tartu linn, Tartu linn, Tartumaa ESTJakobi 2 51005 Tartu linn, Tartu linn, Tartumaa ESTJakobi 2, IV korrus 51005 Tartu linn, Tartu linn, Tartumaa ESTJakobi 2, III korrus, ruumid 302-337 51005 Tartu linn, Tartu linn, Tartumaa ESTÜlikooli 16 51003 Tartu linn, Tartu linn, Tartumaa ESTLossi 3 51003 Tartu linn, Tartu linn, Tartumaa ESTÜlikooli 18 50090 Tartu linn, Tartu linn, Tartumaa ESTPosti 1 71004 Viljandi linn, Viljandimaa ESTJakobi 2 51005 Tartu linn, Tartu linn, Tartumaa ESTJakobi 2 51005 Tartu linn, Tartu linn, Tartumaa ESTSotsiaalteaduste valdkondLossi 36 51003 Tartu linn, Tartu linn, Tartumaa ESTJakobi 5 51005 Tartu linn, Tartu linn, Tartumaa ESTLossi 36, ruum 301 51003 Tartu linn, Tartu linn, Tartumaa ESTNarva mnt 18 51009 Tartu linn, Tartu linn, Tartumaa ESTNäituse 2 50409 Tartu linn, Tartu linn, Tartumaa ESTNäituse 20 - 324 50409 Tartu linn, Tartu linn, Tartumaa ESTLossi 36 51003 Tartu linn, Tartu linn, Tartumaa ESTRaekoja plats 2 20307 Narva linn, Ida-Virumaa ESTRingi 35 80012 Pärnu linn, Pärnu linn, Pärnumaa ESTLossi 36 51003 Tartu linn, Tartu linn, Tartumaa ESTLossi 36 51003 Tartu linn, Tartu linn, Tartumaa ESTMeditsiiniteaduste valdkondRavila 19 50411 Tartu linn, Tartu linn, Tartumaa ESTBiomeedikum, Ravila 19 50411 Tartu linn, Tartu linn, Tartumaa ESTNooruse 1 50411 Tartu linn, Tartu linn, Tartumaa ESTL. Puusepa 1a 50406 Tartu linn, Tartu linn, Tartumaa ESTL. Puusepa 8 50406 Tartu linn, Tartu linn, Tartumaa ESTRavila 19 50411 Tartu linn, Tartu linn, Tartumaa ESTUjula 4 51008 Tartu linn, Tartu linn, Tartumaa ESTRavila 50411 Tartu linn, Tartu linn, Tartumaa ESTRavila 19 50411 Tartu linn, Tartu linn, Tartumaa ESTLoodus- ja täppisteaduste valdkondVanemuise 46 - 208 51003 Tartu linn, Tartu linn, Tartumaa ESTNarva mnt 18 51009 Tartu linn, Tartu linn, Tartumaa ESTRiia 23b/2 51010 Tartu linn, Tartu linn, Tartumaa ESTMäealuse 14 12618 Tallinn, Harjumaa ESTRavila 14a 50411 Tartu linn, Tartu linn, Tartumaa ESTNarva mnt 18 51009 Tartu linn, Tartu linn, Tartumaa ESTRiia 23, 23b - 134 51010 Tartu linn, Tartu linn, Tartumaa ESTObservatooriumi 1 61602 Tõravere alevik, Nõo vald, Tartumaa ESTNooruse 1 50411 Tartu linn, Tartu linn, Tartumaa ESTJ. Liivi tn 2 50409 Tartu linn, Tartu linn, Tartumaa ESTVanemuise 46 51003 Tartu linn, Tartu linn, Tartumaa ESTVanemuise 46 51003 Tartu linn, Tartu linn, Tartumaa ESTNooruse 1 50411 Tartu linn, Tartu linn, Tartumaa ESTAkadeemilise sekretäri tegevusvaldkondLossi 3 51003 Tartu linn, Tartu linn, Tartumaa ESTUppsala 6, Lossi 36 51003 Tartu linn, Tartu linn, Tartumaa ESTFinantsjuhi tegevusvaldkondÜlikooli 17 51005 Tartu linn, Tartu linn, Tartumaa ESTJakobi 4 51005 Tartu linn, Tartu linn, Tartumaa ESTKantsleri tegevusvaldkondUppsala 10 51003 Tartu linn, Tartu linn, Tartumaa ESTÜlikooli 18A (III korrus) 51005 Tartu linn, Tartu linn, Tartumaa ESTÜlikooli 18, ruumid 102, 104, 209, 210 50090 Tartu linn, Tartu linn, Tartumaa ESTArendusprorektori tegevusvaldkondNarva mnt 18 51009 Tartu linn, Tartu linn, Tartumaa ESTVanemuise 46 51003 Tartu linn, Tartu linn, Tartumaa ESTLossi 25 51003 Tartu linn, Tartu linn, Tartumaa ESTRektori tegevusvaldkondÜlikooli 18 50090 Tartu linn, Tartu linn, Tartumaa ESTÕppeprorektori tegevusvaldkondUppsala 10 51003 Tartu linn, Tartu linn, Tartumaa ESTÜlikooli 18b 51005 Tartu linn, Tartu linn, Tartumaa ESTLossi 36-401 51003 Tartu linn, Tartu linn, Tartumaa ESTTeadusprorektori tegevusvaldkondW. Struve 1 50091 Tartu linn, Tartu linn, Tartumaa EST
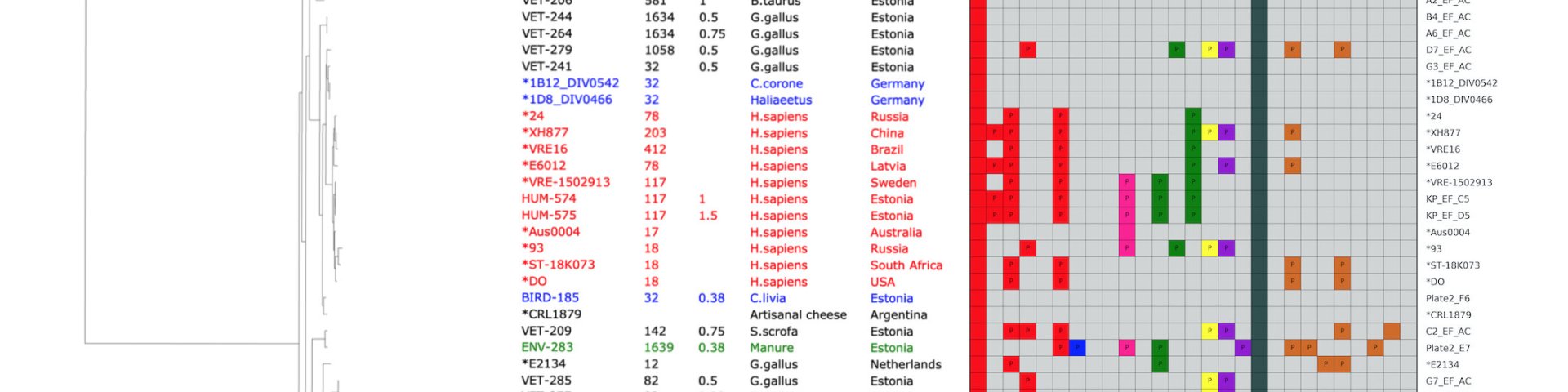
mikroobsete patogeenide DNA tuvastamine inimese verest
haruldaste ja väheuuritud variatsioonide leidmine
toidu koostise ja päritolu määramine toidus leiduva DNA abil
genoomijärjestuste usaldusväärne ja kiire analüüs
Bioinformaatika uurimisgrupp
Meie üldine eesmärk on arendada arvutusalgoritme genoomijärjestuste usaldusväärseks ja kiireks analüüsiks. Mõned näited:
* antimikroobse resistentsuse ennustamine ja plasmiidide tuvastamine bakterikoloonia DNA järjestusest [1,2];
* mikroobsete patogeenide DNA tuvastamine inimese verest (rakuvaba DNA fraktsioonist), reoveest või muudest allikatest;
* haruldaste ja väheuuritud variatsioonide leidmine (Alu-elemendid, endogeensed retroviirused, rRNA geenide koopia arv, jms.) inimeste personaalsetest genoomi järjestustest [3,4];
* toidu koostise ja päritolu määramine toidus leiduva DNA abil [5].
Kaugemas plaanis soovime luua täieliku komplekti tarkvara ja ennustusmudeleid, mis võimaldaksid täpsemalt jälgida meid ümbritsevat keskkonda sh. meid ümbritsevaid patogeene, inimese mikrobioomi ja meie toitu.
- Oselin, K., Kaplinski, L., Jääger, A., Valter, A., Puurand, T. jt. (2023). Comparative Genomic Analysis to Distinguish Triple Metachronous Lung Carcinoma from Metastatic Recurrence: Brief Report on First Case Treated with Three Consecutive VATS Lobectomies. Annals of Medical and Clinical Oncology 5 (145)
- Pajuste, F.D., Remm, M. (2023). GeneToCN: an alignment-free method for gene copy number estimation directly from next-generation sequencing reads. Scientific Reports, 13 (17765)
- Kaplinski, L., Möls, M., Puurand, T., Remm, M. (2023). DOCEST—fast and accurate estimator of human NGS sequencing depth and error rate. Bioinformatics Advances, 3 (1)
- Kaplinski, L., Möls, M., Puurand, T., Pajuste, F.D., Remm, M. (2021). KATK: Fast genotyping of rare variants directly from unmapped sequencing reads. Human Mutation, 42(6):777-786
- Raime, K., Krjutškov, K. Remm, M. (2020). Method for the Identification of Plant DNA in Food Using Alignment-Free Analysis of Sequencing Reads: A Case Study on Lupin. Front. Plant Sci., 11:646
- Puurand, T., Kukuškina, V., Pajuste, F.D., Remm, M. (2019). AluMine: alignment-free method for the discovery of polymorphic Alu element insertions. Mobile DNA, 10:31
- Roosaare, M., Puustusmaa, M., Möls, M., Vaher, M., Remm, M. (2018). PlasmidSeeker: identification of known plasmids from bacterial whole genome sequencing reads. PeerJ, 6: e4588
- Aun, E., Brauer, A., Kisand, V., Tenson, T., Remm, M. (2018). A k-mer-based method for the identification of phenotype-associated genomic biomarkers and predicting phenotypes of sequenced bacteria. PLoS Computational Biology, 14(10):e1006434
Maa Elu - Postimees, 09.01.2025. Mullu nii Eestis kui maailmas laineid löönud mee DNA-testi loonud meeskond töötab nüüd välja metoodikat, et tuvastada meest mesilasi ohustavaid RNA-viiruseid, mis valmistavad peavalu ja majanduslikku kahju mesinikele üle maailma. Artiklis tutvustab projekti lähemalt Celvia CC AS toiduvaldkonna juht Kairi Raime.
25.09.2024 loodus- ja reaalainete õpetajate teaduspäeva programmis osalemine (bioloogilised andmebaasid)
Tartu Ülikooli teaduskooli kaudu läbi viidav bioinformaatika teemaline kursus gümnaasiumiõpilastele (järjepidevalt alates õppeaastast 2021/2022)
Toidu koostise, päritolu ja ohutuse jälgimine toidus oleva DNA järjestamise abil
Ettevõtetega koostöös otsitakse võimalusi veremürgituse kiiremaks diagnoosimiseks


